Научете как да използвате инструменти за OCR, Apache Spark и други компоненти на Apache Hadoop за обработка на PDF изображения в мащаб.
Технологиите за оптично разпознаване на символи (OCR) са напреднали значително през последните 20 години. Въпреки това, през това време е имало малко или никакви усилия за свързване на OCR с разпределени архитектури като Apache Hadoop за обработка на голям брой изображения в почти реално време.
В тази публикация ще научите как да използвате стандартни инструменти с отворен код заедно с компоненти на Hadoop като Apache Spark, Apache Solr и Apache HBase, за да направите точно това за случай на използване на информация за медицинско устройство. По-конкретно, ще използвате публичен набор от данни, за да преобразувате разказния текст в полета за търсене.
Въпреки че този пример се концентрира върху информация за медицински устройства, той може да се приложи в много други сценарии, където се изисква обработка и запазване на изображения. Застрахователните компании, например, могат да направят всичките си сканирани документи във файлове с искове за търсене за по-добро разрешаване на искове. По подобен начин отделът по веригата на доставки в производствено съоръжение може да сканира всички технически листове с данни от доставчици на части и да ги направи достъпни за търсене от анализатори.
Случай на употреба:Регистрация на медицинско устройство
През последните години се наблюдава вълна от промени в областта на електронната регистрация на лекарствени продукти. Стандартът ISO IDMP (Идентификация на медицински продукти) е един такъв формат на съобщения за регистриране на продукти и вещества, съдържащи се в тях, като идентификаторът на лекарствения продукт, ID на опаковката и партидата се използват за проследяване на продуктите в случаи на неблагоприятни преживявания, незаконни внос, фалшифициране и други въпроси на фармакологичната бдителност. Стандартът изисква не само да се регистрират нови продукти, но и че по-старото/архивирано архивиране на всеки продукт, на който публиката може да бъде изложена, трябва също да бъде предоставена в електронен вид.
За да се съобразят със стандартите на IDMP в различни компании, компаниите трябва да могат да изтеглят и обработват данни от множество източници на данни, като RDBMS, както и, в някои случаи, листове с данни за наследени продукти. Въпреки че е добре известно как да се поглъщат данни от RDBMS чрез технологии като Apache Sqoop, обработката на наследени документи изисква малко повече работа. В по-голямата си част документите трябва да бъдат погълнати и съответният текст трябва да бъде програмно извлечен в мащаб, използвайки съществуващите OCR технологии.
Набор от данни
Ще използваме набор от данни от FDA, който съдържа всички 510(k) декларации, подавани някога от производителите на медицински изделия от 1976 г. Раздел 510(k) от Закона за храните, лекарствата и козметиката изисква производителите на устройства, които трябва да се регистрират, да уведомят FDA за намерението им да пуснат на пазара медицинско изделие поне 90 дни предварително.
Този набор от данни е полезен по няколко причини в този случай:
- Данните са безплатни и са обществено достояние.
- Данните се вписват точно в европейския регламент, който влиза в сила през юли 2016 г. (където производителите трябва да спазват новите стандарти за данни). Пълнежите на FDA съдържат важна информация, свързана с извличането на пълна представа за IDMP.
- Форматът на документите (PDF) ни позволява да демонстрираме прости, но ефективни OCR техники, когато работим с документи от множество формати.
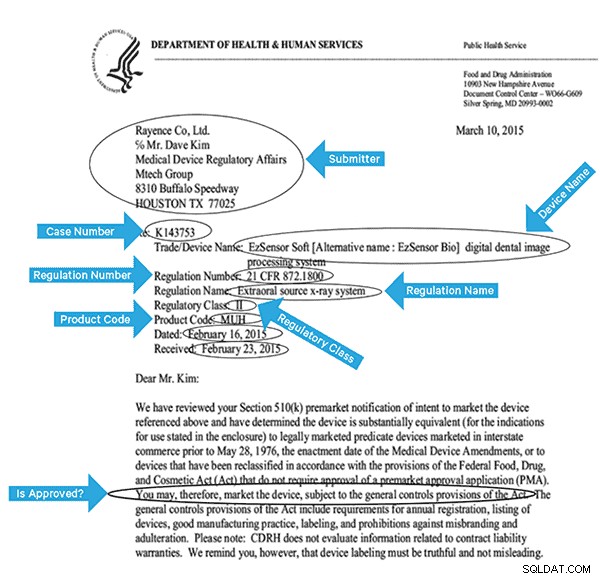
За да индексираме ефективно тези данни, ще трябва да извлечем някои полета от изображенията. По-долу е даден примерен документ с потенциалните полета, които могат да бъдат извлечени.
Архитектура на високо ниво
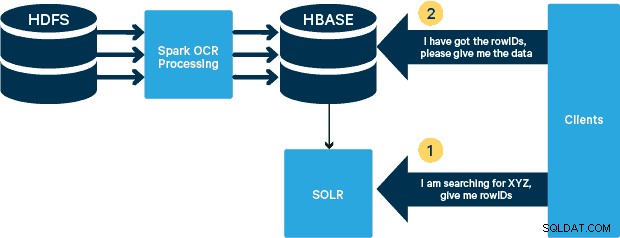
За този случай на употреба PDF файловете се съхраняват в HDFS и се обработват с помощта на библиотеки Spark и OCR. (Стъпката на приемане е извън обхвата на тази публикация, но може да бъде толкова проста, колкото да стартирате hdfs -dfs -put или с помощта на интерфейс webhdfs.) Spark позволява използването на почти идентичен код в приложение Spark Streaming за стрийминг в почти реално време, а HBase е перфектна среда за съхранение на произволен достъп с ниска латентност и е много подходяща за съхранение на изображения, с новата MOB функционалност, за да стартирате. Cloudera Search (което е изградено върху Apache Solr) е единственото решение за търсене, което се интегрира с HBase, като по този начин ви позволява да създавате вторични индекси.
Настройване на таблицата на медицинските устройства в HBase
Ще запазим схемата за нашия случай на употреба ясна. Идентификаторът на реда ще бъде името на файла и ще има две семейства колони:„info“ и „obj“. Семейството на колоните „информация“ ще съдържа всички полета, които извлечехме от изображенията. Семейството на колоните „obj“ ще съдържа байтовете на действителния двоичен обект, в този случай PDF. Името на таблицата в нашия случай ще бъде “mdds.”
Ще се възползваме от функционалността на HBase MOB (среден обект), въведена в HBASE-11339. За да настроите HBase за работа с MOB, са необходими няколко допълнителни стъпки, но удобно инструкциите могат да бъдат намерени на тази връзка.
Има много начини да създадете таблицата в HBase програмно (Java API, REST API или подобен метод). Тук ще използваме обвивката на HBase, за да създадем таблицата „mdds“ (умишлено използвайки описателно име на фамилията на колона, за да направим нещата по-лесни за следване). Искаме фамилията колони „информация“ да се репликира в Solr, но не и данните за MOB.
Командата по-долу ще създаде таблицата и ще активира репликация в семейство колони, наречено „информация“. От решаващо значение е да посочите опцията REPLICATION_SCOPE => '1' , в противен случай HBase Lily Indexer няма да получава никакви актуализации от HBase. Искаме да използваме MOB пътя в HBase за обекти, по-големи от 10MB. За да постигнем това, създаваме и друго семейство колони, наречено „obj“, използвайки следните параметри за MOB:
IS_MOB => вярно, MOB_THRESHOLD => 10240000
IS_MOB параметърът указва дали това семейство колони може да съхранява MOB, докато MOB_THRESHOLD определя след това колко голям трябва да бъде обектът, за да се счита за MOB. И така, нека създадем таблицата:
създайте 'mdds', {NAME => 'информация', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => true, MOB_THRESHOLD => 10240000 /предварително>
За да потвърдите, че таблицата е създадена правилно, изпълнете следната команда в обвивката на HBase:
hbase(main):001:0> describe 'mdds'Таблица mdds е ВКЛЮЧЕНА mddsCOLUMN FAMILIES DESCRIPTION{NAME => 'информация', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'ROW_1'SCOPE => 'ROW_1COPE , VERSIONS => '1', COMPRESSION => 'NONE', MIN_VERSIONS => '0', TTL => 'ЗАВИНАГ', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}{NAME => 'obj', DATA_BLOCK_ENCODING => 'NONE', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '0', COMPRESSION => 'NONE', VERSIONS => '1', MIN_VERSIONS => '0', TTL => 'ЗАВИНАГИ', MOB_THRESHOLD => '10240000', IS_MOB => 'true', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '655536', BLOCKHECHE false', BLOCKHE => 'true'}2 реда(а) за 0,3440 секунди Обработка на сканирани изображения с Tesseract
OCR измина дълъг път по отношение на справянето с вариациите на шрифта, шума на изображението и проблемите с подравняването. Тук ще използваме OCR двигателя с отворен код Tesseract, който първоначално е разработен като собствен софтуер в лабораториите на HP. Оттогава разработката на Tesseract беше пусната като софтуер с отворен код и се спонсорира от Google от 2006 г.
Tesseract е изключително преносима софтуерна библиотека. Той използва библиотеката за обработка на изображения Leptonica, за да генерира двоично изображение, като прави адаптивно прагове на сиво или цветно изображение.
Обработката следва традиционен тръбопровод стъпка по стъпка. Следва грубият ход на стъпките:
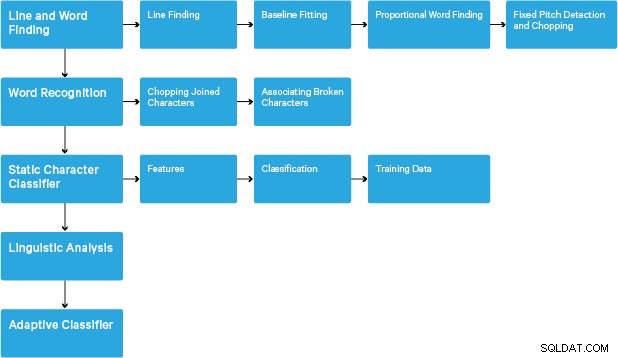
Обработката започва с анализ на свързан компонент, който води до съхраняване на намерените компоненти. Тази стъпка помага при проверка на вложеността на очертанията и броя на очертанията на деца и внуци.
На този етап очертанията се събират, чисто чрез влагане, в двоични големи обекти (BLOB). BLOB-овете са организирани в текстови редове и редовете и регионите се анализират за фиксирана стъпка или пропорционален текст. Текстовите редове се разделят на думи по различен начин според вида на разстоянието между знаците. Текстът с фиксирана стъпка се нарязва незабавно от клетки със знаци. Пропорционалният текст се разделя на думи с помощта на определени интервали и размити интервали.
След това разпознаването протича като процес с два прохода. При първото преминаване се прави опит да се разпознае всяка дума на свой ред. Всяка дума, която е задоволителна, се предава на адаптивен класификатор като данни за обучение. След това адаптивният класификатор получава шанс да разпознае по-точно текст по-надолу в страницата. Тъй като адаптивният класификатор може да е научил нещо полезно твърде късно, за да даде принос близо до горната част на страницата, се извършва второ преминаване през страницата, при което думите, които не са били разпознати достатъчно добре, се разпознават отново. Последната фаза разрешава размити интервали и проверява алтернативни хипотези за височината x, за да намери текст с малки главни букви.
Tesseract в сегашната си форма е напълно съвместим с unicode и е обучен за няколко езика. Въз основа на нашето изследване, това е една от най-точните библиотеки с отворен код, налични за OCR. Както бе споменато по-рано, Tesseract използва Leptonica. Ние също така използваме Ghostscript, за да разделим PDF файловете на изображения. (Можете да разделите във формат за компресиране на изображения по ваш избор; ние избрахме PNG.) Тези три библиотеки са написани на C++ и за да ги извикаме от програми на Java/Scala, трябва да използваме имплементации на съответните интерфейси на Java. В нашата работа използваме JNI връзките от JavaPresets. (Инструкциите за изграждане можете да намерите по-долу.) Използвахме Scala, за да напишем драйвера на Spark.
val renderer :SimpleRenderer =new SimpleRenderer()renderer.setResolution(300)val изображения:List[Image] =renderer.render(document)
Leptonica чете в разделените изображения от предишната стъпка.
ImageIO.write( x.asInstanceOf[RenderedImage], "png", imageByteStream)val pix:PIX =pixReadMem ( ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ), ByteBuffer.wrape(imageByteByteBy). ) ).капацитет( ))
След това използваме извиквания на Tesseract API, за да извлечем текста. Предполагаме, че документите са на английски тук, следователно вторият параметър на метода Init е „eng.“
val api:TessBaseAPI =new TessBaseAPI( )api.Init( null, "eng" )api.SetImage(pix)api.GetUTF8Text().getString()
След като изображенията бъдат обработени, извличаме някои полета от текста и ги изпращаме на HBase.
def populateHbase ( fileName:String, редове:String, pdf:org.apache.spark.input.PortableDataStream) :Unit ={ /** Конфигурирайте и отворете HBase връзка */ val mddsTbl =_conn.getTable( TableName. valueOf( "mdds" )); val cf ="info" val put =new Put( Bytes.toBytes( fileName )) /** * Извличане на полета тук с помощта на регулярни изрази * Създаване на Put обекти и изпращане на HBase */ val aAndCP ="""(?s)(? m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe:(\w\d\d\d\d\d\d).*"" ".r …….. редовете съвпадат { case aAndCP( addr, casenum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "submitter_info" ),Bytes.toBytes( addr ) ).add( Bytes .toBytes( cf ),Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum )) case _ => println( "не съответства на регулярен израз" ) } ……. lines.split("\n").foreach { val regNumRegex ="""Номер на регламента:\s+(.+)""".r val regNameRegex ="""Име на регламент:\s+(.+)""" .r …….. ……. _ съвпадение { case regNumRegex( regNum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "reg_num" ), ……. ….. case _ => print( "" ) } } put.add ( Bytes.toBytes( cf ), Bytes.toBytes( "text" ), Bytes.toBytes( lines )) val pdfBytes =pdf.toArray.clone put.add(Bytes.toBytes( "obj"), Bytes.toBytes( " pdf" ), pdfBytes ) mddsTbl.put( put ) …….}
Ако погледнете отблизо кода по-горе, точно преди да изпратим обекта Put на HBase, ние вмъкваме необработените PDF байтове в семейството на колоните „obj“ на таблицата. Използваме HBase като слой за съхранение на извлечените полета, както и на необработеното изображение. Това го прави бързо и удобно за приложението да извлече оригиналното изображение, ако е необходимо. Пълният код може да бъде намерен тук. (Струва си да се отбележи, че докато използвахме стандартни HBase API за създаване на Put обекти за HBase, в реална производствена система би било разумно да обмислим използването на API на SparkOnHBase, които позволяват пакетни актуализации на HBase от Spark RDD.)
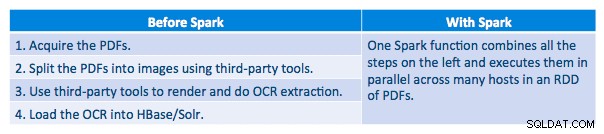
Тръбопровод за изпълнение
Успяхме да обработим всеки PDF в серийна рамка. За да мащабираме обработката, ние избрахме да обработваме тези PDF файлове по разпределен начин с помощта на Spark. Следващата диаграма показва как комбинираме различни етапи от тази обработка, за да превърнем работния процес в просто макро извикване от Spark и да заредим данните в HBase.
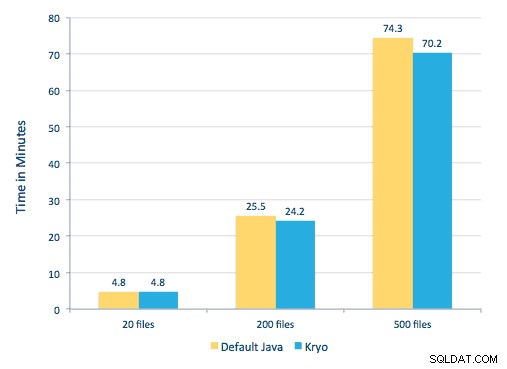
Опитахме се също да направим сравнение между методите за сериализиране, но с нашия набор от данни не видяхме значителна разлика в производителността.
Настройка на средата
Използван хардуер:клъстер с пет възела с 15 GB памет, 4 vCPU и 2x40 GB SSD
Тъй като използвахме C++ библиотеки за обработка, използвахме JNI обвързванията, които могат да бъдат намерени тук.
Създайте JNI връзките за Tesseract и Leptonica от предварително зададени настройки на javaCPP:
-
- На всички възли:
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel
git clone https://github.com/bytedeco/javacpp-presets.git cd javacpp-presets - Създайте Leptonica.
cd leptonica./cppbuild.sh инсталирайте leptonicacd cppbuild/linux-x86_64/leptonica-1.72/LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&sudo make installcd ../../../mvn clean installcd ..
- Създайте Tesseract.
cd tesseract./cppbuild.sh инсталирай tesseractcd tesseract/cppbuild/linux-x86_64/tesseract-3.03LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&make installcd ./ ../../mvn clean installcd ..
- Създайте предварително зададени настройки на javaCPP.
mvn чиста инсталация --projects leptonica,tesseract
Използваме Ghostscript, за да извлечем изображенията от PDF файловете. Инструкциите за изграждане на Ghostscript, съответстващи на използваните тук версии на Tesseract и Leptonica, са както следва. (Уверете се, че Ghostscript не е инсталиран в системата чрез мениджъра на пакети.)
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gztar zxvf ghostscript-9.16.tar.gzcd ghostscript-9.16./autogen.sh &&./configure --prefix=/usr - -disable-compile-inits --enable-dynamicsudo make &&make soinstall &&install -v -m644 base/*.h /usr/include/ghostscript &&ln -v -s ghostscript /usr/include/ps(В зависимост от вашия ldpath настройка, може да се наложи да направите):sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
Уверете се, че всички необходими библиотеки са в пътя на класа. Поставяме всички съответни буркани в директория, наречена lib. Запетаята е важна по-долу:
$ за i в `ls lib/*`; експортирайте MY_JARS=./$i,$MY_JARS; donetesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
Извикваме програмата Spark, както следва. Трябва да посочим extraLibraryPath за собствените библиотеки на Ghostscript; другият conf е необходим за Tesseract.
spark-submit --jars $MY_JARS --num-executors 12 --executor-memory 4G --executor-cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --confspark.executorEnv. TESSDATA_PREFIX=/home/vsingh/javacpp-presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --confspark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/h /lib/htrace-core-3.1.0-inkubating.jar --driver-class-path/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0 -incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer--conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Създаване на колекция Solr
Solr се интегрира доста безпроблемно с HBase чрез Lily HBase Indexer. За да разберете как се извършва интеграцията на интеграцията на Lily Indexer с HBase, можете да прегледате предишната ни публикация в секцията „Разбиране на репликацията на HBase и Lily HBase Indexer“.
По-долу очертаваме стъпките, които трябва да се извършат за създаване на индексите:
- Генерирайте примерен конфигурационен файл schema.xml:
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg
- Редактирайте файла schema.xml в
$HOME/solrcfg , посочвайки полетата, от които се нуждаем за нашата колекция. Пълният файл може да бъде намерен тук.
- Качете конфигурациите на Solr в ZooKeeper:
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg
- Генерирайте колекцията Solr с 2 фрагмента (-s 2) и 2 реплики (-r 2):
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
В командата по-горе създадохме колекция Solr с два параметъра (-s 2) и две реплики (-r 2). Параметрите бяха достатъчни за нашия корпус, но при действително внедряване ще трябва да зададете броя въз основа на други съображения извън нашия обхват на обсъждане тук.
Регистриране на индексатора
Тази стъпка е необходима за добавяне и конфигуриране на индексатора и репликацията на HBase. Командата по-долу ще актуализира ZooKeeper и ще добави mdds_indexer като партньор за репликация за HBase. Той също така ще вмъкне конфигурации в ZooKeeper, които Lily HBase Indexer ще използва, за да посочи правилната колекция в Solr. |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
Аргументи:
-n mdds_indexer– посочва името на индексатора, който ще бъде регистриран в ZooKeeper-c indexer-config.xml– конфигурационен файл, който ще указва поведението на индексатора-cp solr.zk=localhost:2181/solr– определя местоположението на конфигурацията на ZooKeeper и Solr. Това трябва да се актуализира със специфичното за средата местоположение на ZooKeeper.-cp solr.collection=mdds_collection– указва коя колекция да се актуализира. Припомнете си стъпката за конфигуриране на Solr, където създадохме колекция1.
index-config.xml файлът е сравнително лесен в този случай; всичко, което прави, е да посочи на индексатора коя таблица да гледа, класа, който ще се използва като мапер (com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper ) и местоположението на конфигурационния файл Morphline. По подразбиране типът на съпоставяне е зададен на ред , в който случай документът Solr става пълният ред. Param name="morphlineFile" определя местоположението на конфигурационния файл Morphlines. Местоположението може да бъде абсолютен път на вашия Morphlines файл, но тъй като използвате Cloudera Manager, посочете относителния път като morphlines.conf.
Съдържанието на конфигурационния файл на hbase-indexer може да бъде намерено тук.
Конфигуриране и стартиране на Lily HBase Indexer
Когато активирате Lily HBase Indexer, трябва да посочите логиката на трансформацията на Morphlines, която ще позволи на този индексатор да анализира актуализациите на таблицата на медицинските устройства и да извлече всички съответни полета. Отидете на Услуги и изберете Lily HBase Indexer, който сте добавили по-рано. Изберете Configurations->View and Edit->Service-Wide->Morphlines . Копирайте и поставете файла Morphlines.
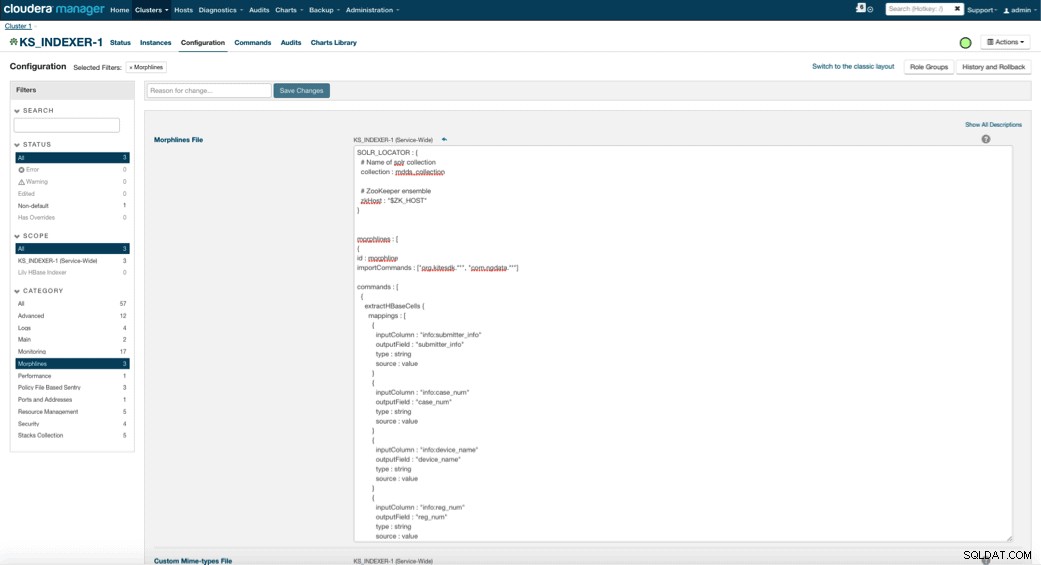
Библиотеката с морфини за медицински устройства ще извърши следните действия:
- Прочетете имейл събитията на HBase с
extractHBaseCellsкоманда - Преобразувайте клеймите за дата/време в поле, което Solr ще разбере, с
convertTimestampкоманди - Изхвърлете всички допълнителни полета, които не сме посочили в schema.xml, с
sanitizeUknownSolrFieldsкоманда
Изтеглете копие на този файл Morphlines от тук.
Една важна забележка е, че полето id ще бъде автоматично генерирано от Lily HBase Indexer. Тази настройка може да се конфигурира във файла index-config.xml по-горе чрез посочване на атрибута unique-key-field. Най-добрата практика е да оставите името по подразбиране на идентификатор – тъй като не е посочено в xml файла по-горе, полето за идентификатор по подразбиране е генерирано и ще бъде комбинация от RowID.
Достъп до данните
Имате избор от много визуални инструменти за достъп до индексираните изображения. HUE и Solr GUI са много добри опции. HBase също така позволява редица техники за достъп, не само от GUI, но и чрез HBase обвивката, API и дори прости техники за скриптове.
Интеграцията със Solr ви дава голяма гъвкавост и може също да предостави много прости, както и разширени опции за търсене на вашите данни. Например, конфигурирането на файла Solr schema.xml така, че всички полета в имейл обекта да се съхраняват в Solr, позволява на потребителите да имат достъп до пълните тела на съобщенията чрез просто търсене, с компромис между пространството за съхранение и сложността на изчисленията. Като алтернатива можете да конфигурирате Solr да съхранява само ограничен брой полета, като идентификатора. С тези елементи потребителите могат бързо да търсят в Solr и да извличат идентификатора на реда, който от своя страна може да се използва за извличане на отделни полета или цялото изображение от самия HBase.
Примерът по-горе съхранява само rowID в Solr, но индексира всички полета, извлечени от изображението. Търсенето в Solr в този сценарий извлича идентификатори на редове в HBase, които след това можете да използвате за запитване на HBase. Този тип настройка е идеална за Solr, тъй като поддържа ниски разходите за съхранение и се възползва напълно от възможностите на Solr за индексиране.
Примерни заявки
По-долу са дадени някои примерни заявки, които могат да бъдат направени от приложението в Solr. Идеята е, че клиентът първоначално ще иска индекси на Solr, връщайки идентификатора на реда от HBase. След това потърсете HBase за останалите полета и/или оригиналното необработено изображение.
- Дайте ми всички документи, подадени между следните дати:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received:[2010-01 -06T23:59:59,999Z ДО 2010-02-06T23:59:59,999Z]
- Дайте ми документи, чиито документи са подадени под регулаторно име за мобилни рентгенови системи:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile рентгенова система
- Дайте ми всички документи, които са подадени от китайски производители:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*Китай*
Идентификаторите от документи на Solr са идентификаторите на редове в HBase; втората част на заявката ще бъде към HBase за извличане на данните (включително необработения PDF, ако е необходимо).
Достъп чрез HUE
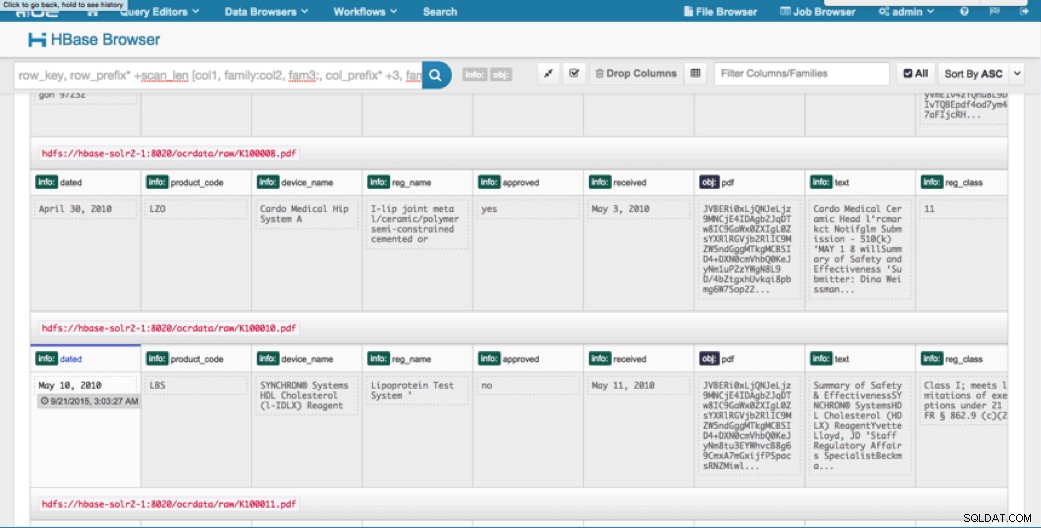
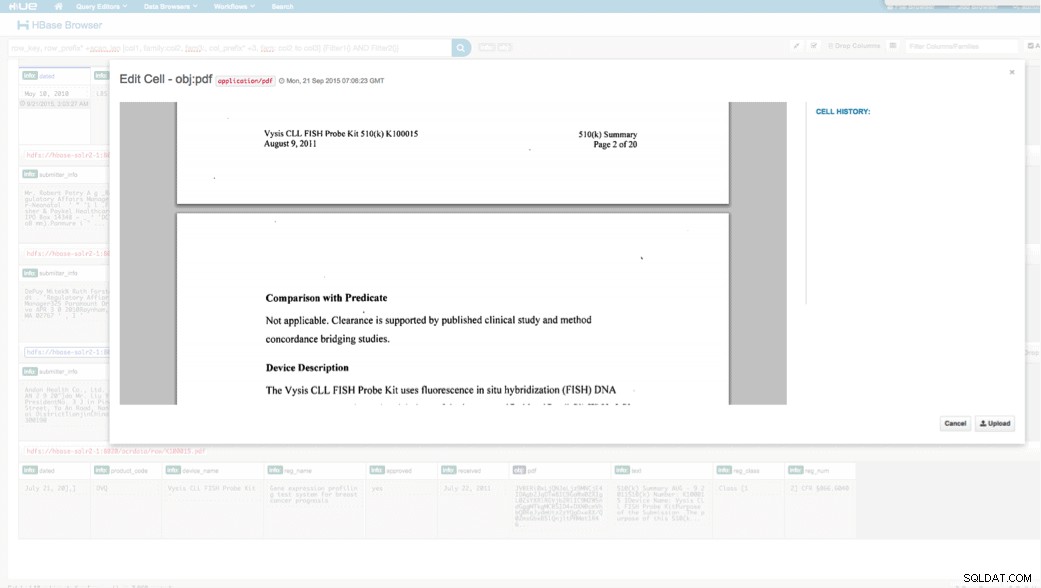
Можем да прегледаме качените данни чрез браузъра HBase в HUE. Едно страхотно нещо за HUE е, че може да открие двоичните файлове за PDF и да ги изобрази, когато щракнете върху.
По-долу е дадена моментна снимка на изгледа на анализираните полета в редовете на HBase, както и визуализиран изглед на един от PDF обектите, съхранени като MOB в семейството на колоните obj.
Заключение
В тази публикация ние демонстрирахме как да използваме стандартни технологии с отворен код за извършване на OCR върху сканирани документи с помощта на мащабируема програма Spark, съхраняване в HBase за бързо извличане и индексиране на извлечената информация в Solr. Трябва да е очевидно, че:
- Като се има предвид формата на спецификацията на съобщението, можем да извлечем полета и двойки стойности и да ги направим достъпни за търсене чрез Solr.
- Тези полета от данни могат да изпълнят изискванията на IDMP да направят наследените данни електронни, което влиза в сила някъде следващата година.
- Полетата, както и необработените изображения, могат да бъдат запазени в HBase и достъпни чрез стандартни API.
Ако откриете, че се нуждаете от обработка на сканирани документи и комбиниране на данните с различни други източници във вашето предприятие, помислете за използването на комбинация от Spark, HBase, Solr, заедно с Tesseract и Leptonica. Това може да ви спести значително време и пари!
Джеф Шмейн е старши архитект на решения в Cloudera. Той има над 16 години опит във финансовата индустрия със силно разбиране на търговията със сигурност, риска и регулациите. През последните няколко години той е работил по различни варианти на използване в 8 от 10 най-големи инвестиционни банки в света.
Вартика Сингх е старши консултант по решения в Cloudera. Тя има над 12 години опит в приложното машинно обучение и разработката на софтуер.